Microbiômica
Serviço completo podendo começar na extração de DNA indo até a bioinformática
para a listagem de microrganismos presentes e comparação das amostras.
GENONE
Metabarcoding 16S, 18S, ITS e outros.
As análises de metagenômica podem incluir abordagens tradicionais de sequenciamento Sanger de 16S para bactérias e arquéias que podem ser cultivadas em laboratório, bem como análises avançadas de comunidades microbianas via regiões variáveis de genes específicos através dos Sequenciamentos de Nova Geração (NGS) de 2ª (short reads) e 3ª geração (long reads), o que é chamado de barcoding. O sequenciamento completo do material genético do microbioma através do sequenciamento shotgun em NGS é uma abordagem frequentemente utilizada para analisar todos os genes presentes em uma comunidade microbiana, fornecendo não apenas análise filogenética, mas também insights sobre as capacidades funcionais de cada espécie dentro da comunidade.
Os genes 16S e 18S rRNA, presentes em procariotos e eucariotos, respectivamente, e o ITS (Internal Transcribed Spacer) em fungos, são considerados marcadores moleculares universais para identificação e análise de comunidades microbianas. Outros genes também podem ser de interesse para caracterização das espécies presentes em determinada condição ou local.
O grupo GenOne oferece o serviço de metabarcoding, através do sequenciamento de regiões variáveis em plataforma Illumina sucesso reconhecido pela comunidade científica brasileira desde 2015.
Contando com o sequenciamento de short reads em plataforma Illumina ou de leituras longas para o sequenciamento completo desses genes em plataforma PacBio, podemos oferecer aos nossos parceiros uma visão mais abrangente da diversidade microbiana em comparação ao sequenciamento de apenas regiões variáveis, superando as limitações impostas nos mais diversos tipos de amostras.
As análises de metagenômica podem incluir abordagens tradicionais de sequenciamento Sanger de 16S para bactérias e arquéias que podem ser cultivadas em laboratório, bem como análises avançadas de comunidades microbianas via regiões variáveis de genes específicos através dos Sequenciamentos de Nova Geração (NGS) de 2ª (short reads) e 3ª geração (long reads), o que é chamado de barcoding. O sequenciamento completo do material genético do microbioma através do sequenciamento shotgun em NGS é uma abordagem frequentemente utilizada para analisar todos os genes presentes em uma comunidade microbiana, fornecendo não apenas análise filogenética, mas também insights sobre as capacidades funcionais de cada espécie dentro da comunidade.
Os genes 16S e 18S rRNA, presentes em procariotos e eucariotos, respectivamente, e o ITS (Internal Transcribed Spacer) em fungos, são considerados marcadores moleculares universais para identificação e análise de comunidades microbianas. Outros genes também podem ser de interesse para caracterização das espécies presentes em determinada condição ou local.
O grupo GenOne oferece o serviço de metabarcoding, através do sequenciamento de regiões variáveis em plataforma Illumina sucesso reconhecido pela comunidade científica brasileira desde 2015.
Contando com o sequenciamento de short reads em plataforma Illumina ou de leituras longas para o sequenciamento completo desses genes em plataforma PacBio, podemos oferecer aos nossos parceiros uma visão mais abrangente da diversidade microbiana em comparação ao sequenciamento de apenas regiões variáveis, superando as limitações impostas nos mais diversos tipos de amostras.

Bioinformática
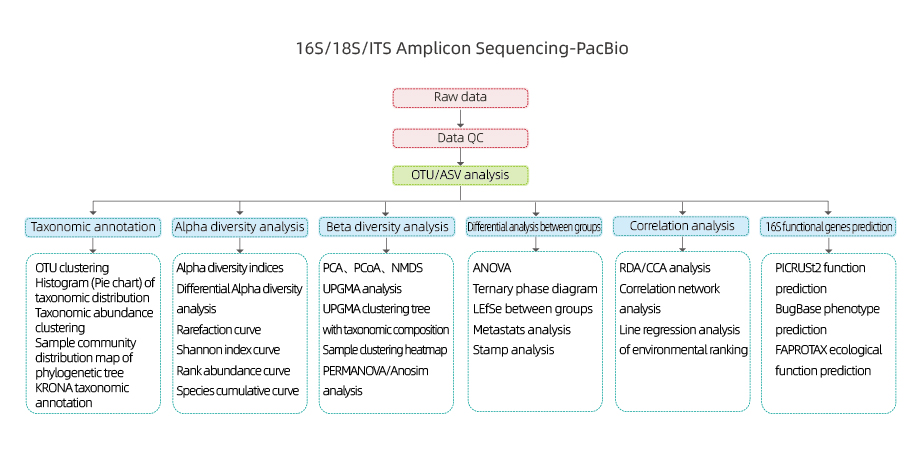
- 1 - Controle de qualidade de dados brutos.
- 2 - Agrupamento de Unidades Taxonômicas Operacionais (OTUs)/ Remoção de ruído (Amplicon Sequence Variants, ASVs).
- 3 - Anotação de OTUs.
- 4 - Diversidade alfa e beta.
- 5 - Análise inter-grupos
- 6 - Identificação de relações entre OTUs e fatores experimentais (por exemplo, pH, temperatura).
- 7- Predição de genes funcionais - Identificação de genes putativos com base em sequências de OTUs ou ASVs (por exemplo, KEGG pathways, CAZymes).
Amostras:
Podemos receber amostras únicas ou até milhares por vez. Só é necessário o envio de DNA purificado, com fragmentos maiores médios maiores que o alvo a ser buscado, concentração superior à 20ng/ul (medida no qubit – nanodrop não é recomendado) e volume de ~20ul. Enviar no mínimo 400ng de DNA por cada alvo Illumina, com fragmentos de DNA acima de 1kb, ou 1ug para PacBio com fragmentos com tamanho médio acima de 2kb (recomendado enviar sempre amostras em concentração e quantidades maiores que as mínimas solicitadas). Quantificar as amostras por fluorescência, eg. Qubit ou similar.
Também podemos receber diversos tipos de amostras para a extração de DNA, como solo, frutas, membranas de filtração, etc. Converse com nossos especialistas.
O controle de qualidade é realizado através da amplificação das amostras com os primers escolhidos. As amostras devem amplificar no tamanho esperado, caso contrário serão recusadas. Tentaremos mudar as condições da PCR para conseguir amplificar a amostra, em cada reação utilizamos cerca de 100-200ng de DNA, por isso se faz necessário o envio de uma quantidade maior. Em caso da presença de inibidores, e se o cliente enviar mais que 1ug de DNA, é possível tentar uma etapa de purificação, valor a combinar.
Podemos receber amostras únicas ou até milhares por vez. Só é necessário o envio de DNA purificado, com fragmentos maiores médios maiores que o alvo a ser buscado, concentração superior à 20ng/ul (medida no qubit – nanodrop não é recomendado) e volume de ~20ul. Enviar no mínimo 400ng de DNA por cada alvo Illumina, com fragmentos de DNA acima de 1kb, ou 1ug para PacBio com fragmentos com tamanho médio acima de 2kb (recomendado enviar sempre amostras em concentração e quantidades maiores que as mínimas solicitadas). Quantificar as amostras por fluorescência, eg. Qubit ou similar.
Também podemos receber diversos tipos de amostras para a extração de DNA, como solo, frutas, membranas de filtração, etc. Converse com nossos especialistas.
O controle de qualidade é realizado através da amplificação das amostras com os primers escolhidos. As amostras devem amplificar no tamanho esperado, caso contrário serão recusadas. Tentaremos mudar as condições da PCR para conseguir amplificar a amostra, em cada reação utilizamos cerca de 100-200ng de DNA, por isso se faz necessário o envio de uma quantidade maior. Em caso da presença de inibidores, e se o cliente enviar mais que 1ug de DNA, é possível tentar uma etapa de purificação, valor a combinar.
Plataforma


Organismos
Bacterias
Fungos
Eucariotos
Genes Funcionais do Ciclo do Nitrogênio
Genes Funcionais do Ciclo do Nitrogênio
Archaeas
Micorrizas arbusculares
Bactéria
Eucariotos
Fungos
Archaeas
Eucariotos
Algas
Eucariotos
Genes Funcionais do Ciclo do Nitrogênio
Região Alvo
Tipo
16SV3+V4
Comum
16S V4+V5
Comum
16S V4
Comum
Endophytes 16S V3+V4
Comum
ITS1
Comum
ITS2
Comum
18S V4
Comum
nirS
Comum
narG
Comum
nosZ
Comum
nifH(nested)
Especial
Ammonia-oxidizing archaea(AOA) amoA
Especial
Ammonia-oxidizing bacteria(AOB) amoB
Especial
nirS
Especial
nirK
Especial
nosZ
Especial
16S V3+V4
Comum
AMF
Especial
16S
Comum
18S
Comum
ITS
Comum
Archaea 16S
Comum
COI
Especial
algae 18S V4
Especial
18S V7
Especial
nifH
Especial
- Primers Comuns: são os mais utilizados na rotina, possuem preços convencionais e geralmente anunciados pela GenOne com tempo de entrega padrão.
- Primers Especiais: são para regiões onde possuímos experiências prévias, porém alguns podem ter baixa eficiência. O custo e prazo para utilização desses primers em geral é superior aos convencionais, consultar cada caso. Em geral necessita de um pedido mínimo de 200 amostras.
- Primers customizados indicados pelos clientes: da mesma forma que os primers especiais o custo e prazo para utilização desses primers é superior aos convencionais.
- Também podemos receber diversos tipos de amostras para a extração de DNA, como solo, frutas, membranas de filtração, etc. Converse com nossos especialistas.
- O controle de qualidade é realizado através da amplificação das amostras com os primers escolhidos.
- As amostras devem amplificar no tamanho esperado, caso contrário serão recusadas.
- Tentaremos mudar as condições da PCR para conseguir amplificar a amostra, em cada reação utilizamos cerca de 100-200ng de DNA, por isso se faz necessário o envio de uma quantidade maior.
- Em caso da presença de inibidores, e se o cliente enviar mais que 1ug de DNA, é possível tentar uma etapa de purificação, valor a combinar.
Fale conosco
Estamos ansiosos para atender
às suas necessidades.
- (21) 3285-9105
- vendas@genone.com.br